

总机:025-58361106-801
传真:025-58361107-806
Email:info@njpeptide.com
地址:南京市化学工业园区方水路158号三楼

近年来,为了应对“禁抗、限抗”给畜牧业带来的挑战,大量科研工作者致力于开发新型抗生素替代物的研究。抗菌肽(antimicrobial peptides,AMPs)广泛存在于动植物和微生物等有机体中,是一类能够帮助机体抵抗外界病原菌的小分子物质[1],同时对多种病原菌、病毒、肿瘤、寄生虫等均具有杀灭作用。其作用机制主要通过物理方式破坏细胞膜完整性, 从而发挥杀菌作用,且不易产生耐药性[2-3],有望成为抗生素替代物之一。有研究表明,在动物饲粮中添加AMPs能够抑制病菌繁殖,有效改善动物肠道菌群结构,提高生产性能[4]。大量的研究已经表明,AMPs在畜牧业上的应用必将为畜牧业的发展带来新的推动力。但由于天然AMPs稳定性低、毒性高以及应用成本较高,很少有成熟的抗菌肽产品应用于畜牧业或临床中[5-6]。因此,优化天然抗菌肽序列,开发具有抗菌、抗生物膜、抗酶解、抗炎、耐盐等功能的抗菌肽成为新的研究热点。近些年,研究者们提出了多种工程肽的设计思路,例如将天然抗菌肽作为模板进行截短或杂合[7-11];再者就是利用结构功能关系或氨基酸间的作用力(例如π-π作用力、氢键等)来构建具有特殊结构和功能的抗菌肽[12-18]。
但是,随着对抗菌药物研究的深入,人们逐渐认识到广谱的抗菌药物在发挥作用的同时也会对非致病菌群造成影响,致使有害菌群的大量定植,引起微生物菌群分布不平衡[19-21],进而引发动物疾病,影响生产性能。同时,长期饲喂广谱抗菌药物极有可能引发多种致病性微生物的抗药性,使得药物对这类细菌感染的预防和治疗效果不佳,造成二次感染和药物残留,进而危害人类健康。因此,高特异性的抗菌肽具有很大的开发潜力。在以往的研究中发现乳酸菌素肽除了其杀伤结构域外,还含有一段靶向结构域,这增加了其对目标细菌的识别和杀灭作用。Eckert等[22]基于这一发现,第一次提出靶向抗菌肽(specifically targeted antimicrobial peptide, STAMP)的概念,在广谱抗菌肽中引入靶向肽段,将其转化为能够选择性杀菌的“智能”化合物。这类“智能”化合物在维持微生物区系稳态和组织细胞健康的同时极大限度地发挥着针对性抗菌作用。在减少药物副作用,保护食品中的益生菌群,保持微生态平衡的基础上实现靶向消除癌细胞、靶向杀灭病原菌,靶向识别感染部位的目的。为此,如何获得高效安全的靶向抗菌肽成为了当下抗菌肽开发的重要研究课题,为使得抗菌肽同时具备抗菌和靶向的双重功能,近年来研究者们从结构功能关系和开发天然抗菌肽等方向出发,赋予抗菌肽主动识别目标菌群的能力,或是利用微环境改变提高抗菌肽活性,设计出一系列具有靶向功能的工程肽,为后人开发新型靶向抗菌肽奠定基础。
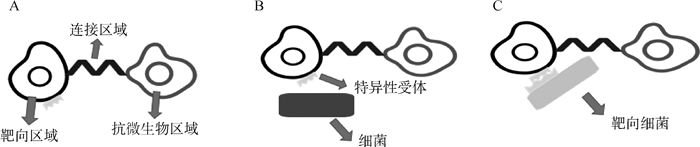
1 融合型靶向抗菌肽的设计融合型靶向抗菌肽可分为三个功能区(如图 1A):靶向结构域(targeting region)、连接结构域(linker region)和杀菌结构域(killing region)。靶向结构域通常能够在微生物菌群中特异性识别目标菌群(如图 1B、C),一般不具有抗菌作用。连接结构域通常由柔性氨基酸,如甘氨酸(glycine,G)和丝氨酸(serine,S)组成,连接两个功能区,避免由于氨基酸的相互作用干扰两个独立的功能区发挥作用。杀菌结构域通常是一条广谱抗菌肽,例如大肠菌素等。
|
A.融合型靶向抗菌肽的组成;B、C.靶向结构域识别目标菌群A.Composition of fusion-targeted antimicrobial peptides; B, C. Targeting region recognizes targeted bacteria图 1 融合型靶向抗菌肽的结构Fig. 1 Structure of fusion specially targeted antimicrobial peptides |
脂多糖(lipopolysaccharide, LPS)仅存在于革兰阴性菌外层细胞膜中[23]。由于LPS中不饱和脂肪酸的存在,阻挡亲脂类抗生素渗透进入磷脂双分子层中[24],容易诱发对诸如利福平、克林霉素、红霉素等亲脂类抗生素的耐药。同时Lázár等[25]通过利用集成方法测定临床分离菌株对不同作用机制的抗菌肽的敏感性,证实外膜上LPS的含量也是影响阳离子抗菌肽发挥作用的主要原因之一。因此LPS被认为是设计抗革兰阴性菌抗菌肽的理想靶标。Muhle和Tam[26]构建半胱氨酸β-折叠框架模拟LPS的结合位点,设计所得的抗菌肽序列及其衍生物对革兰阴性细菌Escherichia coli(E. coil)具有显著的杀菌效果(最小抑菌浓度20 nmol·L-1),抗菌活性高于对革兰阳性细菌Staphylococcus aureus (S. aureus)选择性200倍。同时利用LPS结合基序也能够拓宽抗菌肽的抗菌谱。常见的与LPS有结合作用的蛋白主要有脂多糖结合蛋白(LPS-binding protein, LBP)、杀菌/通透性增加蛋白(bactericidal/permeability-increasing protein, BPI)、血清淀粉样蛋白P(serum amyloid P component, SAP)及中性粒细胞分泌的抗菌蛋白等[27],如表 1所示是LPS结合蛋白中包含的LPS结合肽段。Kim等[28]根据天然的LPS结合基序中氨基酸出现的频率和位置构建了LPS结合肽段,将其连接到抗菌肽GNU7的N端,结果发现LPS结合肽明显提高了GNU7对革兰阴性菌的杀灭活性,拓宽GNU7的抗菌谱。但由于LPS只存在于革兰阴性细菌中,靶向LPS不适用于针对革兰阳性菌靶向药物开发。
|
|
表 1 常见的LPS结合蛋白中LPS结合肽段的氨基酸序列[27-28]Table 1 Comparison of LPS-binding region of common LPS-binding protein[27-28] |
信息素是由多种生物分泌的信号分子。在细菌的世界里,它们不仅能够检测细胞密度并发出信号,还能够与细胞膜上的受体蛋白结合进行信号分子的传递、诱导或抑制信号分子传递到细胞内,调节细菌基因的表达、细菌内酶的释放或毒力因子的产生,调控微生物群体的生理过程,例如生物膜的形成[29-32]。与分泌高丝氨酸内酯的革兰阴性菌不同,革兰阳性菌分泌的信息素多是寡肽。为此,将信息素作为抗菌肽的“靶向”识别区域设计针对革兰阳性菌的抗菌药物具有广阔的应用前景。Qiu等[33]以大肠杆菌素(能够通过破坏细胞膜磷脂双分子层来杀灭大肠杆菌及相关种属细菌)作为杀菌结构域,以金黄色葡萄球菌信息素AgrD1作为靶向结构域,信息素与目标菌特异性受体识别后将细菌素靶向投递至目标菌群中,发挥选择性杀菌的作用。随后Qiu等[34]又将大肠杆菌素与肠球菌分泌的包含7个氨基酸残基的信息素cCf 10进行融合,成功制备了一种针对耐万古霉素粪肠球菌融合靶向抗菌肽。Mao等[35]同样利用金黄色葡萄球菌信息素AgrD1作为靶向区域,与广谱抗菌肽plectasin进行融合,构建针对耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus, MRSA)的靶向抗菌肽。细菌分泌的信息素是一类理想的靶向结构域,将其与广谱抗菌肽融合能够实现对不同细菌靶向杀灭的目的。但革兰阴性菌分泌的高丝氨酸内酯构建靶向抗菌肽具有合成成本高、合成过程复杂等缺点,因此这类方法只适用于设计靶向革兰阳性菌的抗菌肽。
1.3 抗体型抗菌肽的设计抗体类药物是靶向型抗菌药物中的一种,这种药物一直以靶向性和高效性著称,靶向细菌表面侵袭力物质的抗体药物主要是调动宿主免疫系统的补体固定和调理吞噬杀伤(opsonophagocytic killing,OPK)来清除致病菌[36]。例如靶向金黄色葡萄球菌表面抗原的抗体类药物在金黄色葡萄球菌和表皮葡萄球菌细胞壁锚定蛋白的IgG1(rF1)和靶向金黄色葡萄球菌自溶酶亚基氨基葡萄糖苷酶的单抗的基础上,设计的靶向金黄色葡萄球菌抗体药物[37];Horn等[38]则开发了一种靶向铜绿假单胞菌表面抗原的抗体药物,其靶点是O11血清型铜绿假单胞菌脂多糖。该单抗在体外表现出OPK活性并能够减少细菌感染数量,增加感染模型中动物的存活率。Touti等[39]利用靶向识别的单克隆抗体与抗菌肽相结合,设计合成了一种靶向E. coil的抗体-抗菌肽结合物,实现了靶向与杀菌功能的结合。但由于这种蛋白类药物的生产成本高,化学合成效率低以及稳定性低等问题,使其很难应用于生产中。Eckert等[22]则对单克隆抗体肽段进行筛选,筛选出靶向铜绿假单胞菌(Pseudomonas spp)的短肽,将其与广谱抗菌肽novispirin G10结合,设计出靶向铜绿假单胞菌的小分子肽类药物。虽然现有的抗菌抗体药物还有一定的限制条件,例如蛋白类药物容易引起机体免疫原性反应,但是抗菌抗体药物可能在不久的将来改变预防和治疗细菌感染的整体格局。
1.4 噬菌体肽库筛选型抗菌肽的设计噬菌体展示技术(phage display)是将外源基因整合到噬菌体核酸中,表达的外源蛋白质与噬菌体的衣壳蛋白融合,再利用噬菌体筛选技术将具有特异性识别功能的多肽筛选出来[40]。噬菌体展示技术已被广泛应用于蛋白质的研究中,用于开发多肽和蛋白质药物,利用蛋白质之间的相互作用筛选具有靶向功能的多肽等[41]。Anandakumar等[42]构建随机肽噬菌体库,筛选特异性靶向白色念珠菌(Candida albicans,C. albicans)的肽段。McCarthy等[43]突破了噬菌体展示技术只能呈现由天然氨基酸组成的肽的限制,在噬菌体展示多肽中引入2-乙酰苯基硼酸,诱导肽段与细菌细胞表面的动态共价结合,筛选对临床耐药金黄色葡萄球菌和鲍曼不动杆菌具有高度特异性的结合序列。因此,噬菌体展示技术可以用于设计靶向抗菌肽,但这类抗菌肽的靶向机制尚不明确。
融合型靶向抗菌肽的设计思路是将靶向肽段与杀菌肽段进行融合,使其各自发挥功能,这类抗菌肽一般肽链相对较长,成本较高。
2 根据抗菌肽的结构功能参数设计靶向抗菌肽大多数靶向抗菌肽通常是在广谱抗菌肽的N端或C端引入一段具有靶向功能的肽段,通过靶向肽段与目标菌受体蛋白识别,将杀菌肽段投递至目标菌细胞膜表面发挥靶向抗菌作用。但氨基酸之间的相互作用可能会改变肽链的结构,使得靶向功能或者杀菌功能减弱甚至丧失。因此,研究者们开始从抗菌肽的结构功能关系出发设计靶向抗菌肽。
抗菌肽的结构功能参数(包括两亲性、疏水性、正电荷数、二级结构以及氨基酸的组成)对抗菌肽的生物学活性起决定性的作用。Mishra等[44]对抗菌肽数据库(the Antimicrobial Peptide Database,APD)中3 014条天然抗菌肽序列进行比对后发现,靶向革兰阳性菌的抗菌肽一般具有低阳离子性和高疏水性的特性,而靶向革兰阴性菌的抗菌肽一般是具有低疏水性、高阳离子性的特性,部分序列还富含脯氨酸。因此,调整抗菌肽的结构功能参数能够直接影响抗菌肽的抗菌谱。Zhu等[45]通过对螺旋肽RI16的疏水面中心位置氨基酸进行替换,获得具有靶向铜绿假单胞菌的抗菌肽序列;Wang等[13]用柔性配对的甘氨酸(glycine,Gly)替代序列中的丙氨酸(alanine,Ala),也获得了对革兰阴性菌具有较好杀伤效果的窄谱肽。Shang等[46]用色氨酸(tryptophan, Trp)替代蛙皮肤抗菌肽temporin-1CE中不同位置的疏水性氨基酸,结果表明Trp位置改变能够影响抗菌肽与LPS的结合能力,影响抗菌谱。
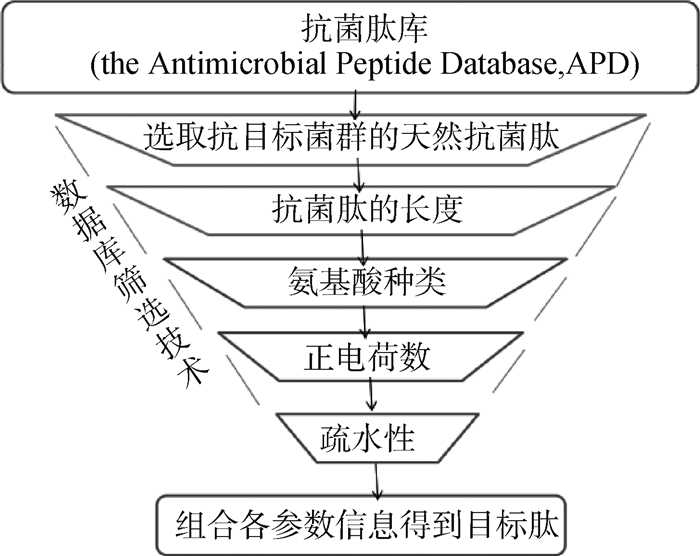
上述方法通过单个氨基酸的替换实现了靶向的目的,但这类靶向抗菌肽的设计方法并不系统。Mishra和Wang[47]利用APD更加系统地分析天然抗菌肽库中抗金黄色葡萄球菌抗菌肽的基本结构参数,根据肽链长度、总电荷数和疏水性等关键参数进行分类,筛选出概率最大的参数值,根据这些参数进行靶向金黄色葡萄球菌抗菌肽的设计。Chou等[48]将APD筛选技术与抗菌肽模板相结合,利用APD筛选出的氨基酸,根据对称结构原则进行排列,同时引入能够与细菌细胞膜发生强烈相互作用的Trp,成功构建一系列靶向革兰阴性菌的窄谱肽。抗菌肽库筛选策略保留了天然抗菌肽中杀灭某种特定菌株应具备的结构参数,更有针对性地进行正电荷和疏水性的排布,与其他设计方法相比成功的概率较大(图 2)。
|
图 2 数据库筛选技术Fig. 2 Database filtering technology |
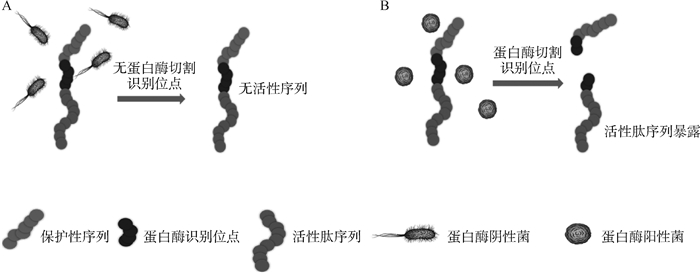
酶触前体药物活性释放系统(directed enzyme prodrug therapy, DEPT)是在靶细胞或是靶器官中利用酶活性局部激活前体药物,释放药物活性[49]。Li等[50]研制了一种新型细菌感染诊断探针(PPA-PLGVRG-Van),包括信号分子(pyropheophorbide-α, PPA)、酶应答肽连接体(明胶酶酶切位点PLGVRG)和靶向配体(vancomycin, Van),Van与革兰阳性菌细胞壁通过多重氢键进行结合,阳性菌过度表达明胶酶选择性地切断PLGVRG连接体,减少了信号分子PPA分子的空间位阻,引发疏水结构域PPA自组装,超分子聚集体能有效地增强光声信号,以实现对细菌感染部位的实时成像,准确识别并判断细菌感染部位。Wang等[51]将明胶酶识别序列作为连接体,连接抗菌肽HHC36生物相容性肽RGD,构建具有细菌选择性的生物材料,这种设计策略降低了HHC36的细胞毒性,同时赋予其生物选择性,为今后制备智能多功能材料提供理论基础(图 3)。Qi等[52]同样利用明胶酶识别序列开发了一种壳聚糖肽。在细菌感染微环境中,阳性菌分泌明胶酶切断明胶酶识别位点,剥离聚乙二醇(polyethylene glycol, PEG)保护层,破坏疏水/亲水平衡。通过壳聚糖的链相互作用,自发地促进纳米粒子重组为纤维结构发挥抗菌作用。利用酶触反应激发抗菌肽分子抗菌活性是设计靶向抗菌肽的新型策略,其有效地克服了传统靶向抗菌肽设计方法的弊端,同时这种策略也可以有效地应用于设计检测细菌感染等新型高分子材料中,具有不错的开发前景。
|
A.蛋白酶阴性菌未分泌蛋白酶,前体肽中和活性中心的抗菌活性;B.蛋白酶阳性菌分泌蛋白酶,酶切前体肽,活性中心暴露发挥抗菌作用A.No protease was secreted by protease-negative bacteria, and pre-peptides decrease the antibacterial activity of active center; B. Protease-positive bacteria secrete proteases, which cleave pre-peptides, and the peptide shows the targeted antimicrobial activity图 3 蛋白酶激活性抗菌肽的作用机制[51]Fig. 3 Mechanism of protease-activated AMPs[51] |
另一个能够影响抗菌肽活性的环境因素是pH。机体内存在许多酸性环境,例如口腔、阴道、创伤口等。pH的改变能够引起抗菌肽结构发生改变从而影响抗菌活性。Xiong等[53]将谷氨酸(阴离子基)和叔胺(阳离子基)交替排列,利用酸性环境下谷氨酸质子化的特性,使得带有正电荷的叔胺在胃中酸性环境下聚合形成α螺旋,杀灭幽门螺旋杆菌(Helicobacter pylori,H. pylori),克服了临床中质子泵抑制剂(proton pump inhibitor, PPI)与抗生素联合治疗带来的副作用。Song等[54]同样利用谷氨酸(glutamate, Glu)在酸性环境下质子化的特性打破Glu和赖氨酸(lysine, Lys)之间的静电吸引靶向杀灭肿瘤细胞。这种pH靶向策略不同于传统的肿瘤治疗药物,能够大大降低对正常组织细胞的毒副作用。另外,富含组氨酸(histidine, His)的抗菌肽在低pH环境中也能表现出良好的抗菌活性。His是一种碱性氨基酸,但由于其等电点较低,正电性小于赖氨酸和精氨酸。通常在酸性环境下才表现出正电性。自然界中存在许多富含His的小肽类物质,如凝血酶衍生的C-末端肽,能够在皮肤局部酸性炎症环境中抑制细菌生长,加速伤口愈合[55]。一些研究者们认为低pH环境中组氨酸肽正电性增加,增加其与阴离子细菌表面之间静电吸引力,实现pH调控活性的目的(图 4)[56]。然而,Li等[57]用苯丙氨酸(phenylalanine, Phe)和His设计了一系列酸激活的抗菌肽(acid-activated peptides, AAPs),结果发现,低pH环境下组氨酸肽抗菌活性增强不仅仅是由于细菌细胞膜的静电吸引力的增强,而是由于干扰质子转运蛋白而发挥抗菌作用。Dennison等[58]和Lai等[59]用His和异亮氨酸(isoleucine, Ile)开发了一系列抗酶解活性肽,结果发现在酸性环境中靶向革兰阴性细菌,他们推测这种靶向机制可能是由于革兰阳性菌膜上的pH依赖性变化引起的。
4 靶向抗菌肽的应用靶向抗菌肽在延续了广谱抗菌肽低耐药性的同时增强了抗细菌感染的靶向性,使得靶向抗菌肽在临床和畜牧生产中表现出了巨大的应用前景。众所周知,现阶段通常依靠杀菌漱口水和口服广谱抗生素来治疗龋齿,但是广谱的抗菌药物能够破坏整个口腔细菌微生态平衡,使得变异链球菌(Streptococcus mutans,S. mutan)和共生细菌之间平等竞争,导致S. mutan在牙齿表面大量定植,感染口腔,形成龋齿。因此,开发针对S. mutan的靶向抗菌药物能够在消除S. mutan的同时建立健康的口腔生物系统,能够长期有效地保护牙齿免受龋齿威胁[19]。在畜牧生产中,哺乳动物的肠道内存在着大量以细菌为主的共生微生物群落。这些肠道菌群能够促进肠道组织发育,如双歧杆菌、乳酸菌等有益菌合成动物机体生长发育所必需的维生素;促进动物机体对多种离子的吸收;为宿主提供多种动物机体自身不具备的酶与生化反应的底物;分解动物机体不易消化的多糖、寡聚糖以及糖蛋白质等物质[60-62]。但畜牧生产中抗生素的滥用,使得肠道内对药物敏感的有益菌群受到不同程度的威胁。另外,当肠道正常菌群受到抗生素的影响后,天然免疫中的重要模式识别受体Toll样受体基因表达水平下降,其下游的适应性免疫功能降低,影响外周免疫器官的正常功能,从而导致机体免疫功能下降[63]。同时母体抗生素的使用也会破坏子代建立正常肠道菌群,造成肠道微生态紊乱,菌群失衡。
开发靶向抗菌肽替代抗生素作为饲料添加剂,能够在维持体内菌群稳态的同时有效发挥抗菌作用,提高生产性能。“精准”靶向已经为疾病治疗和药物开发提供新的思路。而多肽类药物为靶向抗菌药物的研究与开发提供了新的起点。靶向抗菌肽在未来有望成为药物的有效传递载体,例如用于鉴定细菌感染或是靶向消除肿瘤的药物,促进药物浓集于靶器官、靶组织、靶细胞或细胞内结构,避免药物在非特异性健康组织中积聚,实现靶向治疗、靶向杀灭的目的[64]。本文总结了近年来靶向抗菌药物的设计思路,以期在更多疾病的靶向治疗中发挥更大的作用,为将来靶向抗菌肽的开发与设计提供思路。
| [1] | FJELL C D, HISS J A, HANCOCK R E W, et al. Designing antimicrobial peptides:form follows function[J]. Nat Rev Drug Discov, 2012, 11(1): 37–51. DOI: 10.1038/nrd3653 |
| [2] |
单安山, 田昊天, 邵长轩, 等. 抗菌肽抗细菌机理研究进展[J]. 东北农业大学学报, 2018, 49(3): 84–94. SAN A S, TIAN H T, SHAO C X, et al. Research advance on antibacterial mechanism of antimicrobial peptides[J].Journal of Northeast Agricultural University, 2018, 49(3): 84–94. DOI: 10.3969/j.issn.1005-9369.2018.03.010 (in Chinese) |
| [3] | TRAVKOVA O G, MOEHWALD H, BREZESINSKI G. The interaction of antimicrobial peptides with membranes[J]. Adv Colloid Interface Sci, 2017, 247: 521–532. DOI: 10.1016/j.cis.2017.06.001 |
| [4] | YOON J H, INGALE S L, KIM J S, et al. Effects of dietary supplementation of synthetic antimicrobial peptide-A3 and P5 on growth performance, apparent total tract digestibility of nutrients, fecal and intestinal microflora and intestinal morphology in weanling pigs[J]. Livest Sci, 2014, 159: 53–60. DOI: 10.1016/j.livsci.2013.10.025 |
| [5] | BROGDEN N K, BROGDEN K A. Will new generations of modified antimicrobial peptides improve their potential as pharmaceuticals?[J]. Int J Antimicrob Agents, 2011, 38(3): 217–225. DOI: 10.1016/j.ijantimicag.2011.05.004 |
| [6] | WANG J J, DOU X J, SONG J, et al. Antimicrobial peptides:Promising alternatives in the post feeding antibiotic era[J]. Med Res Rev, 2019, 39(3): 831–859. DOI: 10.1002/med.21542 |
| [7] | LV Y F, WANG J J, GAO H, et al. Antimicrobial properties and membrane-active mechanism of a potential α-helical antimicrobial derived from cathelicidin PMAP-36[J]. PLoS One, 2014, 9(1): e86364. DOI: 10.1371/journal.pone.0086364 |
| [8] | YANG Z Y, HE S Q, WANG J J, et al. Rational design of short peptide variants by using Kunitzin-RE, an amphibian-derived bioactivity peptide, for acquired potent broad-spectrum antimicrobial and improved therapeutic potential of commensalism coinfection of pathogens[J]. J Med Chem, 2019, 62(9): 4586–4605. DOI: 10.1021/acs.jmedchem.9b00149 |
| [9] | XU W, ZHU X, TAN T T, et al. Design of embedded-hybrid antimicrobial peptides with enhanced cell selectivity and anti-biofilm activity[J]. PLoS One, 2014, 9(6): e98935. DOI: 10.1371/journal.pone.0098935 |
| [10] | HE J, ANDERSON M H, SHI W Y, et al. Design and activity of a 'dual-targeted' antimicrobial peptide[J]. Int J Antimicrob Agents, 2009, 33(6): 532–537. DOI: 10.1016/j.ijantimicag.2008.11.013 |
| [11] | DONG N, CHOU S L, LI J W, et al. Short symmetric-end antimicrobial peptides centered on β-turn amino acids unit improve selectivity and stability[J]. Front Microbiol, 2018, 9: 2832. DOI: 10.3389/fmicb.2018.02832 |
| [12] | CHOU S L, SHAO C X, WANG J J, et al. Short, multiple-stranded β-hairpin peptides have antimicrobial potency with high selectivity and salt resistance[J]. Acta Biomater, 2016, 30: 78–93. DOI: 10.1016/j.actbio.2015.11.002 |
| [13] | WANG J J, CHOU S L, XU L, et al. High specific selectivity and membrane-active mechanism of the synthetic centrosymmetric α-helical peptides with Gly-Gly pairs[J]. Sci Rep, 2015, 5(1): 15963. DOI: 10.1038/srep15963 |
| [14] | SHAO C X, TIAN H T, WANG T Y, et al. Central β-turn increases the cell selectivity of imperfectly amphipathic α-helical peptides[J]. Acta Biomater, 2018, 69: 243–255. DOI: 10.1016/j.actbio.2018.01.009 |
| [15] | XU L, CHOU S L, WANG J J, et al. Antimicrobial activity and membrane-active mechanism of tryptophan zipper-like β-hairpin antimicrobial peptides[J]. Amino Acids, 2015, 47(11): 2385–2397. DOI: 10.1007/s00726-015-2029-7 |
| [16] | WANG J J, CHOU S L, YANG Z Y, et al. Combating drug-resistant fungi with novel imperfectly amphipathic palindromic peptides[J]. J Med Chem, 2018, 61(9): 3889–3907. DOI: 10.1021/acs.jmedchem.7b01729 |
| [17] | WANG J J, SONG J, YANG Z Y, et al. Antimicrobial peptides with high proteolytic resistance for combating gram-negative bacteria[J]. J Med Chem, 2019, 62(5): 2286–2304. DOI: 10.1021/acs.jmedchem.8b01348 |
| [18] | ZHU X, DONG N, WANG Z Y, et al. Design of imperfectly amphipathic α-helical antimicrobial peptides with enhanced cell selectivity[J]. Acta Biomater, 2014, 10(1): 244–257. |
| [19] | GUO L H, EDLUND A. Targeted antimicrobial peptides:a novel technology to eradicate harmful Streptococcus mutans[J]. J Calif Dent Assoc, 2017, 45(10): 557–564. |
| [20] | ECKERT R, HE J, YARBROUGH D K, et al. Targeted killing of Streptococcus mutans by a pheromone-guided "smart" antimicrobial peptide[J]. Antimicrob Agents Chemother, 2006, 50(11): 3651–3657. DOI: 10.1128/AAC.00622-06 |
| [21] | HAN J H, BILKER W B, NACHAMKIN I, et al. Impact of antibiotic use during hospitalization on the development of gastrointestinal colonization with Escherichia coli with reduced fluoroquinolone susceptibility[J]. Infect Control Hosp Epidemiol, 2013, 34(10): 1070–1076. DOI: 10.1086/673155 |
| [22] | ECKERT R, QI F X, YARBROUGH D K, et al. Adding selectivity to antimicrobial peptides:rational design of a multidomain peptide against Pseudomonas spp[J]. Antimicrob Agents Chemother, 2006, 50(4): 1480–1488. DOI: 10.1128/AAC.50.4.1480-1488.2006 |
| [23] |
杨颜铱, 陈芸, 高爽, 等. 抗菌肽抑制脂多糖诱导的炎症反应[J]. 动物营养学报, 2016, 28(12): 3770–3776. YNG Y Y, CHEN Y, GAO S, et al. Inhibitory effects of antimicrobial peptides on lipopolysaccharide-induced inflammation[J]. Chinese Journal of Animal Nutrition, 2016, 28(12): 3770–3776. (in Chinese) |
| [24] | NIKAIDO H. Preventing drug access to targets:cell surface permeability barriers and active efflux in bacteria[J].Semin Cell Dev Biol, 2001, 12(3): 215–223. DOI: 10.1006/scdb.2000.0247 |
| [25] | LÁZÁR V, MARTINS A, SPOHN R, et al. Antibiotic-resistant bacteria show widespread collateral sensitivity to antimicrobial peptides[J]. Nat Microbiol, 2018, 3(6): 718–731. DOI: 10.1038/s41564-018-0164-0 |
| [26] | MUHLE S A, TAM J P. Design of gram-negative selective antimicrobial peptides[J]. Am Chem Soc, 2001, 40(19): 5777–5785. |
| [27] | DE HAAS C J, VAN DER ZEE R, BENAISSA-TROUW B, et al. Lipopolysaccharide (LPS)-binding synthetic peptides derived from serum amyloid P component neutralize LPS[J]. Infect Immun, 1999, 67(6): 2790–2796. DOI: 10.1128/IAI.67.6.2790-2796.1999 |
| [28] | KIM H, JANG J H, KIM S C, et al. Enhancement of the antimicrobial activity and selectivity of GNU7 against Gram-negative bacteria by fusion with LPS-targeting peptide[J]. Peptides, 2016, 82: 60–66. DOI: 10.1016/j.peptides.2016.05.010 |
| [29] | HWANG I Y, LEE H L, HUANG J G, et al. Engineering microbes for targeted strikes against human pathogens[J]. Cell Mol Life Sci, 2018, 75(15): 2719–2733. DOI: 10.1007/s00018-018-2827-7 |
| [30] | HUO L J, HUANG X Y, LING J Q, et al. Selective activities of STAMPs against Streptococcus mutans[J]. Exp Ther Med, 2018, 15(2): 1886–1893. |
| [31] | KAUFMANN G F, PARK J, JANDA K D. Bacterial quorum sensing:a new target for anti-infective immunotherapy[J].Expert Opin Biol Ther, 2008, 8(6): 719–724. DOI: 10.1517/14712598.8.6.719 |
| [32] | EVEN-TOV E, BENDORI S O, VALASTYAN J, et al. Social evolution selects for redundancy in bacterial quorum sensing[J]. PLoS Biol, 2016, 14(2): e1002386. DOI: 10.1371/journal.pbio.1002386 |
| [33] | QIU X Q, WANG H, LU X F, et al. An engineered multidomain bactericidal peptide as a model for targeted antibiotics against specific bacteria[J]. Nat Biotechnol, 2003, 21(12): 1480–1485. DOI: 10.1038/nbt913 |
| [34] | QIU X Q, ZHANG J, WANG H, et al. A novel engineered peptide, a narrow-spectrum antibiotic, is effective against vancomycin-resistant Enterococcus faecalis[J]. Antimicrob Agents Chemother, 2005, 49(3): 1184–1189. DOI: 10.1128/AAC.49.3.1184-1189.2005 |
| [35] | MAO R Y, TENG D, WANG X M, et al. Design, expression, and characterization of a novel targeted plectasin against methicillin-resistant Staphylococcus aureus[J]. Appl Microbiol Biotechnol, 2013, 97(9): 3991–4002. DOI: 10.1007/s00253-012-4508-z |
| [36] |
王志明. 抗生素研发新方向:抗菌抗体药物[J]. 中国新药杂志, 2016, 25(19): 2199–2204. WNG Z M. The new direction in the development of antibiotics:anti-bacterial antibody drugs[J]. Chinese Journal of New Drugs, 2016, 25(19): 2199–2204. (in Chinese) |
| [37] | HAZENBOS W L W, KAJIHARA K K, VANDLEN R, et al. Novel staphylococcal glycosyltransferases SdgA and SdgB mediate immunogenicity and protection of virulence-associated cell wall proteins[J]. PLoS Pathog, 2013, 9(10): e1003653. DOI: 10.1371/journal.ppat.1003653 |
| [38] | HORN M P, ZUERCHER A W, IMBODEN M A, et al. Preclinical in vitro and in vivo characterization of the fully human monoclonal IgM antibody KBPA101 specific for Pseudomonas aeruginosa serotype IATS-O11[J]. Antimicrob Agents Chemother, 2010, 54(6): 2338–2344. |
| [39] | TOUTI F, LAUTRETTE G, JOHNSON K D, et al. Antibody-bactericidal macrocyclic peptide conjugates to target gram-negative bacteria[J]. Chembiochem, 2018, 19(19): 2039–2044. DOI: 10.1002/cbic.201800295 |
| [40] | SOROKULOVA I B, OLSEN E V, CHEN I H, et al. Landscape phage probes for Salmonella typhimurium[J]. J Microbiol Methods, 2005, 63(1): 55–72. |
| [41] | HUANG J X, BISHOP-HURLEY S L, COOPER M A. Development of anti-infectives using phage display:biological agents against bacteria, viruses, and parasites[J]. Antimicrob Agents Chemother, 2012, 56(9): 4569–4582. DOI: 10.1128/AAC.00567-12 |
| [42] | ANANDAKUMAR S, BOOSI K N, BUGATHA H, et al. Phage displayed short peptides against cells of Candida albicansdemonstrate presence of species, morphology and region specific carbohydrate epitopes[J]. PLoS One, 2011, 6(2): e16868. DOI: 10.1371/journal.pone.0016868 |
| [43] | MCCARTHY K A, KELLY M A, LI K C, et al. Phage display of dynamic covalent binding motifs enables facile development of targeted antibiotics[J]. J Am Chem Soc, 2018, 140(19): 6137–6145. DOI: 10.1021/jacs.8b02461 |
| [44] | MISHRA B, NARAYANA J L, LUSHNIKOVA T, et al. Low cationicity is important for systemic in vivo efficacy of database-derived peptides against drug-resistant Gram-positive pathogens[J]. Proc Natl Acad Sci U S A, 2019, 116(27): 13517–13522. DOI: 10.1073/pnas.1821410116 |
| [45] | ZHU X, SHAN A S, MA Z, et al. Bactericidal efficiency and modes of action of the novel antimicrobial peptide T9W against Pseudomonas aeruginosa[J]. Antimicrob Agents Chemother, 2015, 59(6): 3008–3017. DOI: 10.1128/AAC.04830-14 |
| [46] | SHANG D J, ZHANG Q, DONG W B, et al. The effects of LPS on the activity of Trp-containing antimicrobial peptides against Gram-negative bacteria and endotoxin neutralization[J]. Acta Biomater, 2016, 33: 153–165. DOI: 10.1016/j.actbio.2016.01.019 |
| [47] | MISHRA B, WANG G S. Ab initio design of potent anti-MRSA peptides based on database filtering technology[J]. J Am Chem Soc, 2012, 134(30): 12426–12429. DOI: 10.1021/ja305644e |
| [48] | CHOU S L, WANG J J, SHANG L, et al. Short, symmetric-helical peptides have narrow-spectrum activity with low resistance potential and high selectivity[J]. Biomater Sci, 2019, 7(6): 2394–2409. DOI: 10.1039/C9BM00044E |
| [49] | MIZUKAMI S, KASHIBE M, MATSUMOTO K, et al. Enzyme-triggered compound release using functionalized antimicrobial peptide derivatives[J]. Chem Sci, 2017, 8(4): 3047–3053. DOI: 10.1039/C6SC04435B |
| [50] | LI L L, MA H L, QI G B, et al. Pathological-condition-driven construction of supramolecular nanoassemblies for bacterial infection detection[J]. Adv Mater, 2016, 28(2): 254–262. DOI: 10.1002/adma.201503437 |
| [51] | WANG L, CHEN J J, ZENG X Z, et al. Mechanistic insights and rational design of a versatile surface with cells/bacteria recognition capability via orientated fusion peptides[J]. Adv Sci, 2019, 6(9): 1801827. DOI: 10.1002/advs.201801827 |
| [52] | QI G B, ZHANG D, LIU F H, et al. An "On-Site Transformation" strategy for treatment of bacterial infection[J]. Adv Mater, 2017, 29(36): 1703461. DOI: 10.1002/adma.201703461 |
| [53] | XIONG M H, BAO Y, XU X, et al. Selective killing of Helicobacter pylori with pH-responsive helix-coil conformation transitionable antimicrobial polypeptides[J]. Proc Natl Acad Sci U S A, 2017, 114(48): 12675–12680. DOI: 10.1073/pnas.1710408114 |
| [54] | SONG J J, ZHANG W, KAI M, et al. Design of an acid-activated antimicrobial peptide for tumor therapy[J]. Mol Pharm, 2013, 10(8): 2934–2941. DOI: 10.1021/mp400052s |
| [55] | HOLDBROOK D A, SINGH S, CHOONG Y K, et al. Influence of pH on the activity of thrombin-derived antimicrobial peptides[J]. Biochim Biophys Acta Biomembr, 2018, 1860(11): 2374–2384. DOI: 10.1016/j.bbamem.2018.06.002 |
| [56] | MAISETTA G, VITALI A, SCORCIAPINO M A, et al. pH-dependent disruption of Escherichia coli ATCC 25922 and model membranes by the human antimicrobial peptides hepcidin 20 and 25[J]. FEBS J, 2013, 280(12): 2842–2854. DOI: 10.1111/febs.12288 |
| [57] | LI L N, HE J, ECKERT R, et al. Design and characterization of an acid-activated antimicrobial peptide[J]. Chem Biol Drug Des, 2010, 75(1): 127–132. DOI: 10.1111/j.1747-0285.2009.00904.x |
| [58] | DENNISON S R, MORTON L H, HARRIS F, et al. Low pH enhances the action of maximin H5 against staphylococcus aureus and helps mediate lysylated phosphatidylglycerol-induced resistance[J]. Biochemistry, 2016, 55(27): 3735–3751. DOI: 10.1021/acs.biochem.6b00101 |
| [59] | LAI Z H, TAN P, ZHU Y J, et al. Highly Stabilized α-helical coiled coils kill gram-negative bacteria by multicomplementary mechanisms under acidic condition[J]. ACS Appl Mater Interfaces, 2019, 11(25): 22113–22128. DOI: 10.1021/acsami.9b04654 |
| [60] | DAI Z L, WU G, ZHU W Y. Amino acid metabolism in intestinal bacteria:links between gut ecology and host health[J]. Front Biosci (Landmark Ed), 2011, 16: 1768–1786. DOI: 10.2741/3820 |
| [61] |
姜东京, 张丽, 曹雨诞, 等. 肠道菌群在中药研究中的应用[J]. 中国中药杂志, 2016, 41(17): 3218–3225. JANG D J, ZHANG L, CAO Y D, et al. Application of gut microbiota in research of Chinese medicines[J]. China Journal of Chinese Materia Medica, 2016, 41(17): 3218–3225. (in Chinese) |
| [62] | SOLDAVINI J, KAUNITZ J D. Pathobiology and potential therapeutic value of intestinal short-chain fatty acids in gut inflammation and obesity[J]. Dig Dis Sci, 2013, 58(10): 2756–2766. DOI: 10.1007/s10620-013-2744-4 |
| [63] |
杨泽冉, 辛毅, 侯洁, 等. 肠道菌群失调及其相关疾病研究进展[J]. 山东医药, 2016, 56(1): 99–101. YNG Z R, XIN Y, HOU J, et al. Advances in research on intestinal flora imbalance and related diseases[J]. Shandong Medicine, 2016, 56(1): 99–101. DOI: 10.3969/j.issn.1002-266X.2016.01.039 (in Chinese) |
| [64] | SONG N N, ZHAO L Z, ZHU M L, et al. Recent progress in LyP-1-based strategies for targeted imaging and therapy[J]. Drug Deliv, 2019, 26(1): 363–375. DOI: 10.1080/10717544.2019.1587047 |